You are here
Multiphysics Biofluidics Simulation
マルチフィジックス血流動力学シミュレーション
Massimo Bernaschi (IAC, National Resource Counsil, Italy)
松岡聡 (東京工業大学学術国際情報センター)
背景と概要
動脈硬化をはじめとする心臓血管の疾病を深く理解するには,血液循環を詳細に理解する必要があります.しかし詳細に現実的なサイズのシミュレーションを行うためには,物理モデリングの分野においても高性能計算の分野においても解決するべき課題があります.たとえば現実的な血流動力学のモデルは,血管の複雑な形状に対応できる必要があり,その形状は心拍の影響でも変化します.そして血しょうなどの流体と,赤血球(RBC)・白血球をはじめとする粒子の両方を扱える必要があります.過去の流体・粒子双方のシミュレーションは毛細血管などを対象としており,心臓血管などに対応するにはより多くの計算資源が必要となります.この研究では,人間の心臓血管を対象としたマルチスケールシミュレーションを,東工大TSUBAME2.0スパコンの4,000GPUを用いて行いました.解像度は5cmから10μmの範囲で,約10億ノードの流体と約3億粒子の赤血球の計算を行います.

赤血球は楕円球として扱われる
シミュレーション方法
上述のマルチフィジックス・マルチスケールシミュレーションは,著者らによるMUPHYプログラムによって行います.MUPHYは流体のためのLattice Boltzmann (LB)法と,本目的に特化した分子動力学法(MD)のコードからなり,結合したシミュレーションが可能となります.そしてGPUを用いた実験のために,CUDA版も実装されました.この実装の流体部分では,GPUが主要な演算を行っている間にCPUがノード間境界部分の通信を行うという,計算・通信オーバラップがなされており,効率的な多数GPU演算を可能としています.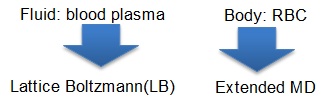
シミュレーションの対象領域は複雑な形状のため,この領域を4000GPUの間で効率的に分割するのも課題となります.本実験では,既存のソフトウェアであるPT-SCOTCHを用いました.これはグラフ分割ツールであるSCOTCHの並列版です.
実験結果
TSUBAME2.0のNVIDIA Tesla M2050 GPUsを4000枚用いたシミュレーションの性能について示します.TSUBAME2.0の計算ノードはGPUを3枚持つため,1,334ノード用いました.シミュレーションは下記を対象とします:
- 流体においては10億のlattice sites
- 3億粒子の赤血球
原則的に計算は単精度にて行われます.システムソフトウェアとしては,CUDAコンパイラ3.2,Intel Fortranコンパイラ11.1,OpenMPI 1.4.2が用いられました.
全体の速度性能としては600TFlops (=0.6PFlops)が得られました.このときの並列化効率は90%以上であり,LB部分については著者らの以前の実験の2倍以上の性能を達成しました.この実験から,TSUBAME2.0全体を8時間利用すると,マイクロ秒の解像度で一心拍全体のシミュレーションが可能となることが分かりました.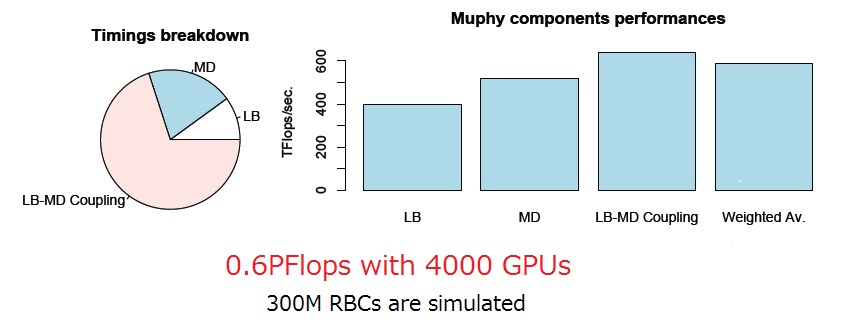
参考文献
M. Bernaschi, M. Bisson, T.Endo, M. Fatica, S. Matsuoka, S. Melchionna, S. Succi. Petaflop biofluidics simulations on a two million-core system. In Proceedings of IEEE/ACM Supercomputing ’11, Seattle, 2011. (To appear)
![[GSIC]東京工業大学学術国際情報センター(学際大規模情報基盤共同利用・共同研究拠点)](../_img/logo.jpg)